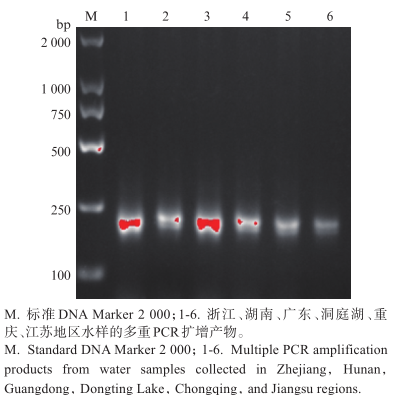
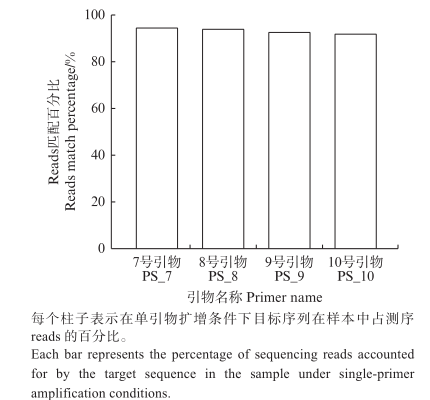
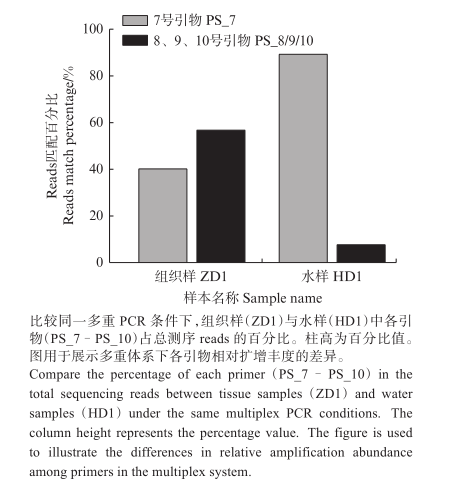
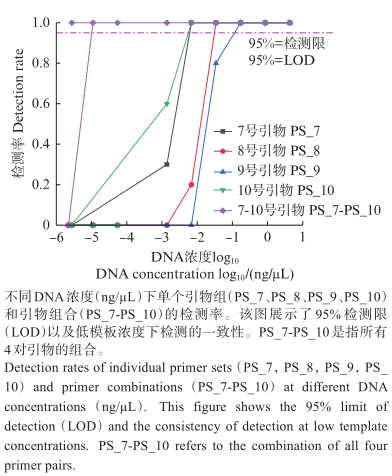
基于环境DNA多重PCR技术的大薸入侵监测引物开发与验证
发布日期:2025-11-03
浏览次数:10次
文章信息/
外来物种入侵是全球生态安全主要威胁之一,每年造成全球超1万亿美元损失。
大薸作为我国淡水生态系统中危害严重的外来入侵植物,最初从巴西引进作为青绿饲料,后因水域环境变化(水位上升、水质恶化、氮磷富集),在云南、广西等南方省份淡水河道及库区快速扩散,抑制本地生物繁殖,威胁生态安全。
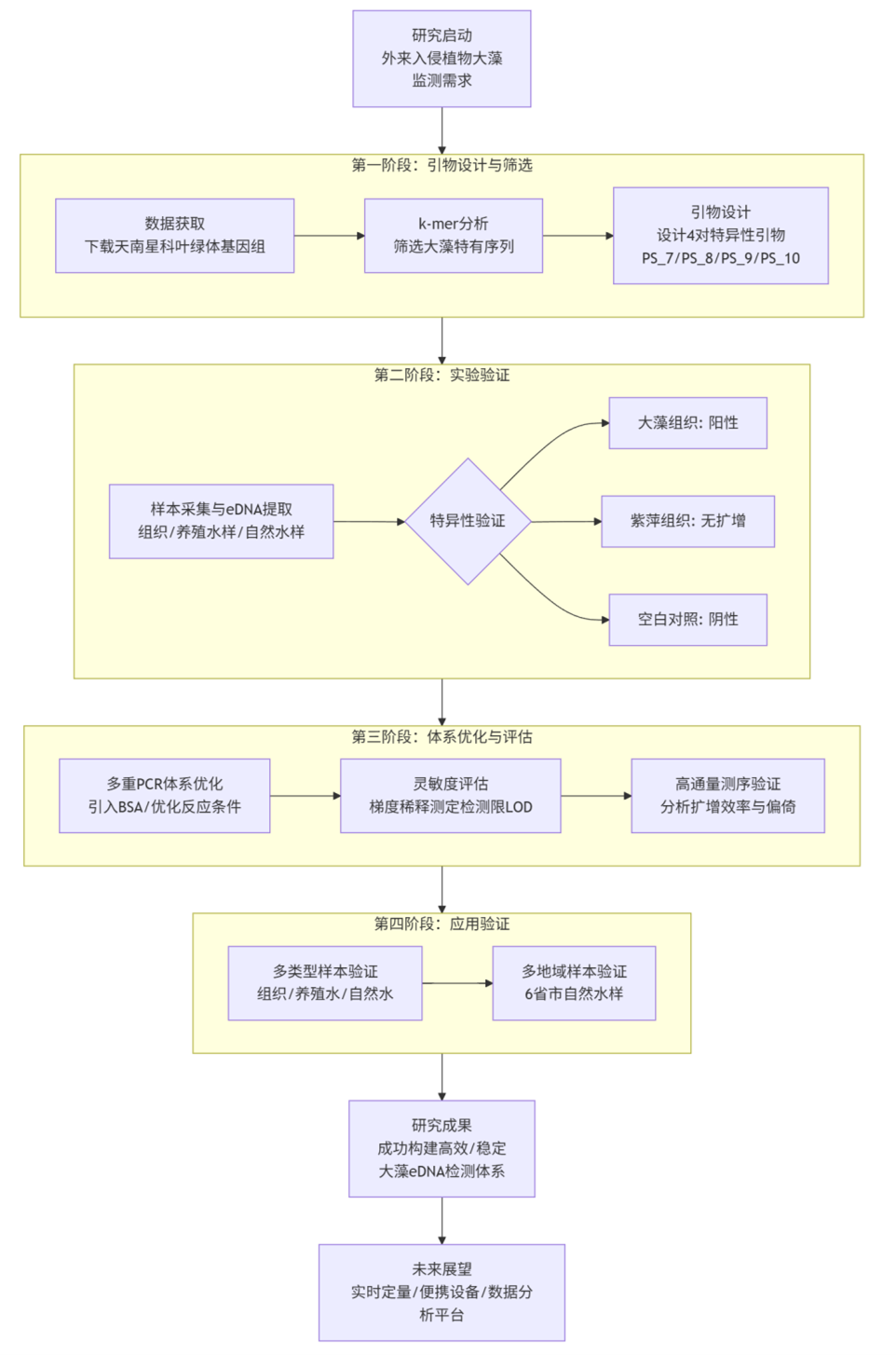
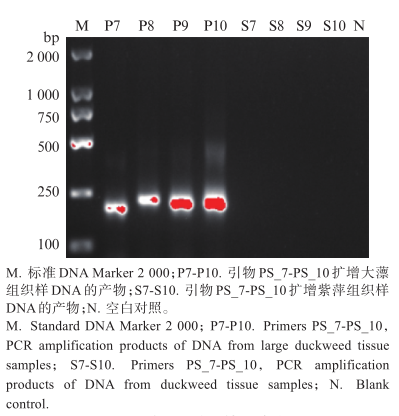
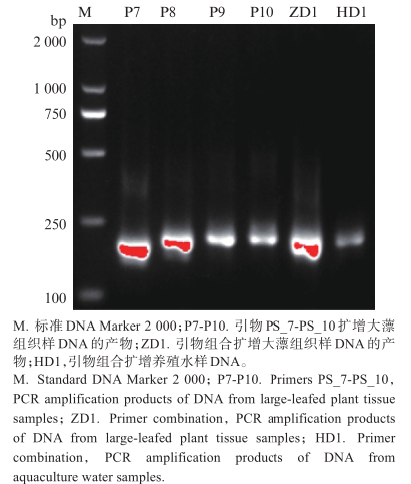
传统形态学调查难以发现入侵早期零散分布的大薸个体或碎片,延误防控时机。环境DNA(eDNA)技术虽在入侵物种监测中具优势,但水生植物eDNA因释放机制特殊(依赖根系分泌、组织脱落等被动释放)、易降解,检测灵敏度和特异性面临挑战,且现有研究多依赖有限基因片段设计引物,缺乏近缘物种全叶绿体基因组比较。因此,开发高灵敏度、高特异性的大薸eDNA检测方法,对其早期预警和防控至关重要。 文章亮点/ -- 实验过程实拍 -- 创新技术路线 首次将k-mer基因组特征分析与叶绿体全基因组比对结合,用于设计水生植物eDNA引物。 高灵敏度多重PCR体系 构建4对特异性引物,检测限低至1.1×10⁻⁵ ng/µL,比单引物提升625倍。 广泛适用性验证 在组织样本、养殖水样和自然水体中均表现稳定,适用于复杂环境。 高效检测无交叉 整个检测流程可在3小时内完成,适合野外快速监测。与近缘物种(如紫萍)无扩增交叉,特异性强。 样本照片 研究结果/ 图1 技术路线 1 引物设计与筛选结果 2 特异性测试结果 图 2 四种引物特异性测试结果 Fig.2 Specificity test results of four primer sets 3 多重 PCR 体系优化 图 3 单个引物与4个引物组合扩增结果 Fig.3 Amplification results of single primer and combination of 4 primers 图 4 多重引物组扩增自然水样结果 Fig.4 Amplification results of multiplex primer sets for natural water samples 4 测序结果 图 5 单引物在单对PCR中的检测效果 Fig.5 Performance of single-primer sets in singleplex PCR 图 6 多重PCR中不同引物在组织与水样中的扩增比例比较 Fig.6 Comparison of read ratios amplified with different primer sets in multiplex PCR between tissue and water samples 5 灵敏度评估结果 图 7 单个引物以及组合引物灵敏度测试结果 Fig.7 Sensitivity test results for single primers and primer combination 总结报告 / zongjie